Naukowcy z Zakładu Mikrobiologii UMB - kierowanego przez dr hab. Katarzynę Leszczyńską -
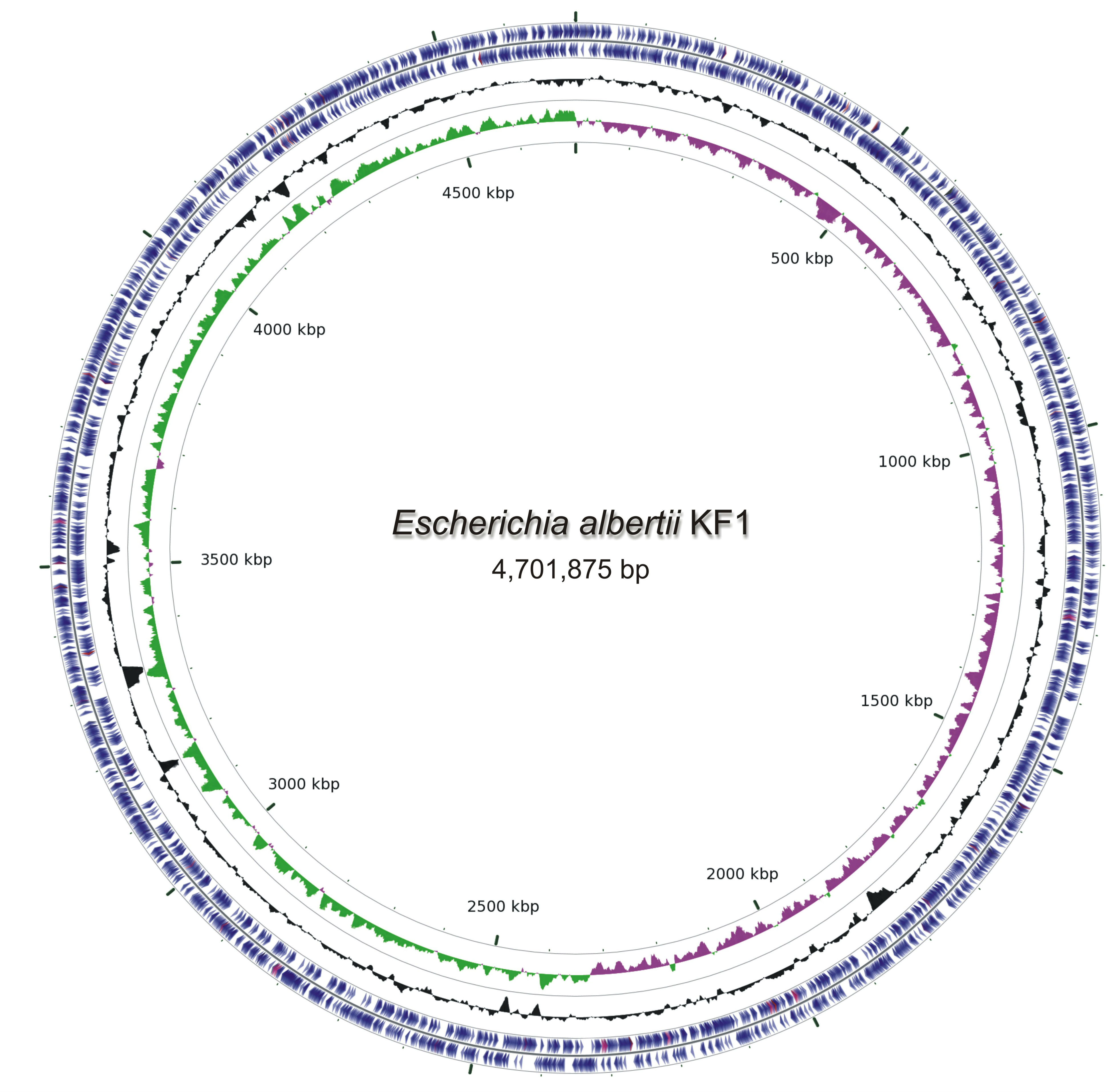
jako pierwsi na świecie złożyli pełną sekwencję genomu bakterii Escherichia albertii.
Biegunka(diarrhoea) jest jedną z najczęstszych przyczyn umieralności dzieci poniżej 5 roku życia i to niezależnie od regionu geograficznego. Problem wysokiej śmiertelności powodowanej biegunkami dotyczy przede wszystkim krajów rozwijających się, natomiast w krajach uprzemysłowionych jest przyczyną ogromnych obciążeń natury socjoekonomicznej.
Należy zaznaczyć, że szczegółowa wiedza o drobnoustrojach wywołujących biegunki jest nadal niepełna. Ze względu na dużą liczbę enteropatogenów stanowi duże wyzwanie diagnostyczne. Do tej pory zidentyfikowano ponad 20 różnych bakterii, wirusów i pasożytów, którym udowodniono lub wykazano potencjalny związek z biegunkami. Dzięki zastosowaniu nowoczesnych metod diagnostycznych, takich jak techniki biologii molekularnej lista ta jest sukcesywnie wydłużana. Nadal jednak w wielu przypadkach nie udaje się wykazać potencjalnego czynnika etiologicznego biegunek.
Escherichia albertii jest słabo poznaną bakterią, ale zapis genetyczny świadczy, że to potencjalny enteropatogen, mający wiele cech wspólnych ze szczepami E. coliwywołującymi biegunki. Historia związku tych bakterii z zakażeniami przewodu pokarmowego sięga końca lat 90. ubiegłego wieku, kiedy to zostały uznane za prawdopodobną przyczynę biegunki u piątki dzieci z Bangladeszu, jednak początkowo były mylone z innymi bakteriami tzn. z Hafnia alvei. Szczegółowa analiza tych szczepów wykazała jednak znaczne różnice na poziomie fenotypowym i genetypowym w porównaniu do referencyjnych szczepów H. alvei. Na podstawie dalszych badań m.in. w oparciu o analizę 16S rRNA stwierdzono, że szczepy te największe pokrewieństwo wykazują z rodzajami Escherichia i Shigella.Ostatecznie zakwalifikowano je do rodzaju Escherichia, a obecną nazwę Escherichia albertii nadano dopiero w 2003 r.
Należy zaznaczyć, że komercyjne systemy do identyfikacji bakterii stosowane powszechnie w laboratoriach mikrobiologicznych nie rozpoznają lub błędnie klasyfikują E. albertii.Obecnie najbardziej wiarygodną metodą identyfikacji E. albertii jest wykrycie specyficznych sekwencji DNA metodą PCR. Ponieważ szczepy E. albertii posiadają geneae, wykorzystywany jako jeden z markerów przy identyfikacji enterokrwotocznych (EHEC) i enteropatogennych E. coli (EPEC), do niedawna część z nich była błędnie identyfikowana.
Intensywne badania nad E. albertii rozpoczęliśmy wiosną 2013 roku, współpraca z zespołem dr hab. Izabeli Święcickiej, prof. UwB, kierownikiem Zakładu Mikrobiologii Uniwersytetu w Białymstoku oraz przychylność władz uczelni i pracowników działu nauki umożliwiła zespołowi Zakładu Mikrobiologii UMB do końca roku złożyć w całość genom tej bakterii. Nadal jednak nie wiadomo, jak często bakterie te występują w próbkach klinicznych, jakie jest ich źródło oraz czy wszystkie szczepy mogą być przyczyną biegunek.
Na częstość występowania i względną proporcję każdego z enteropatogenów ma wpływ wiele różnych czynników. Należy ponadto zaznaczyć, że wraz z upływem czasu występowanie poszczególnych enteropatogenów może się zmieniać, dlatego też sprawne monitorowanie sytuacji epidemiologicznej w danej populacji wymaga stałej aktualizacji danych na temat potencjalnych czynników etiologicznych, częstości ich występowania oraz dróg szerzenia. Ponadto, połączenie danych epidemiologicznych i klinicznych może zostać wykorzystane do oceny stopnia ryzyka wystąpienia biegunek o określonej etiologii w zależności od różnych zmiennych oraz opracowania modeli pomocnych klinicystom w podjęciu decyzji jaką drogę diagnostyczną powinni obrać.
Jak istotny jest to problem przekonano się w 2011 roku w Niemczech podczas występowania zakażenia epidemicznym szczepem E. coli EHEC O104:H4, który charakteryzował się typowymi dla gatunku właściwościami biochemicznymi, co uniemożliwiało odróżnienie go w hodowli od fizjologicznych pałeczek okrężnicy. Dopiero szczegółowa charakterystyka szczepu EHEC O104:H4, który wywołał ognisko w Niemczech i innych krajach, wykazała, że był to szczep o bardzo unikalnych właściwościach, dlatego też ognisko również miało nietypowy, nieobserwowany do tej pory przebieg.Zjadliwość tego hiperwirulentnego szczepu była wypadkową czynników zjadliwości charakterystycznych dla prototypowych szczepów zaliczanych do enterokrwotocznych (EHEC) oraz enteroagregacyjnych E. coli (EAEC). Izolat ten charakteryzował się również unikatowym profilem lekowrażliwości, należał do wielolekoopornych. Cecha ta była związana z nabyciem plazmidu kodującego β-laktamazę CTX-M15, obecnego w genomie wielu innych pałeczek Enterobacteriaceae, lecz dotychczas niespotykanego u EHEC.
Zaistniała sytuacja potwierdza, że w każdej chwili możemy mieć do czynienia z nietypowym patogenem, który ze względu na swoje właściwości chorobotwórcze będzie stanowić ogromne zagrożenie dla zdrowia publicznego oraz dodatkowo wykazała, że rutynowa diagnostyka mikrobiologiczna w kierunku zakażeń enteropatogenami powinna ulec istotnej modyfikacji. Natomiast trzeba podkreślić, iż działania naukowców z różnych krajów, umożliwiły prawie natychmiastowe zidentyfikowanie czynnika zakaźnego i przygotowanie procedury jego identyfikacji, co potwierdza bardzo dobrą naukową międzynarodową współpracę w zakresie wykrywania i zwalczania infekcji przenoszonych przez żywność i wodę (tzw. foodborne and waterborne diseases) w krajach UE.
W przypadku problemów z identyfikacją atypowych enteropatogenów pracownicy Zakładu Mikrobiologii UMB gotowi są pomóc w ich diagnostyce, wystarczy tylko skontaktować się (mikrob@umb.edu.pl) i przesłać izolat.
Dr hab. Katarzyna Leszczyńska,
kierownik Zakładu Mikrobiologii UMB





